目的:利用MNE实现自定义矩阵大脑拓扑图的绘制
0、加载python库
1
2
3
4
5
6
| import numpy as np
import matplotlib.pyplot as plt
import pandas as pd
import mne
%matplotlib qt
|
1、获取可用的电极布局系统
在使用MNE进行EEG信号的可视化操作时,往往需要导入对应电极的位置信息,MNE中有内置的常见电极布局系统,通过调用下面指令进行导入:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
| mne.channels.get_builtin_montages()
# output:
['standard_1005',
'standard_1020',
'standard_alphabetic',
'standard_postfixed',
'standard_prefixed',
'standard_primed',
'biosemi16',
'biosemi32',
'biosemi64',
'biosemi128',
'biosemi160',
'biosemi256',
'easycap-M1',
'easycap-M10',
'EGI_256',
'GSN-HydroCel-32',
'GSN-HydroCel-64_1.0',
'GSN-HydroCel-65_1.0',
'GSN-HydroCel-128',
'GSN-HydroCel-129',
'GSN-HydroCel-256',
'GSN-HydroCel-257',
'mgh60',
'mgh70',
'artinis-octamon',
'artinis-brite23',
'brainproducts-RNP-BA-128']
|
从上面的输出可以看出,MNE中共有27个可用的电极布局系统,具体选择哪一个要看你采集数据时使用的脑电帽电极布局系统是哪个。
2、利用MNE自带的电极布局系统对矩阵进行通道定位
2.1 加载脑地形图位置坐标并可视化
1
2
3
| int32_montage = mne.channels.make_standard_montage('biosemi32')
int32_montage.plot()
plt.show()
|
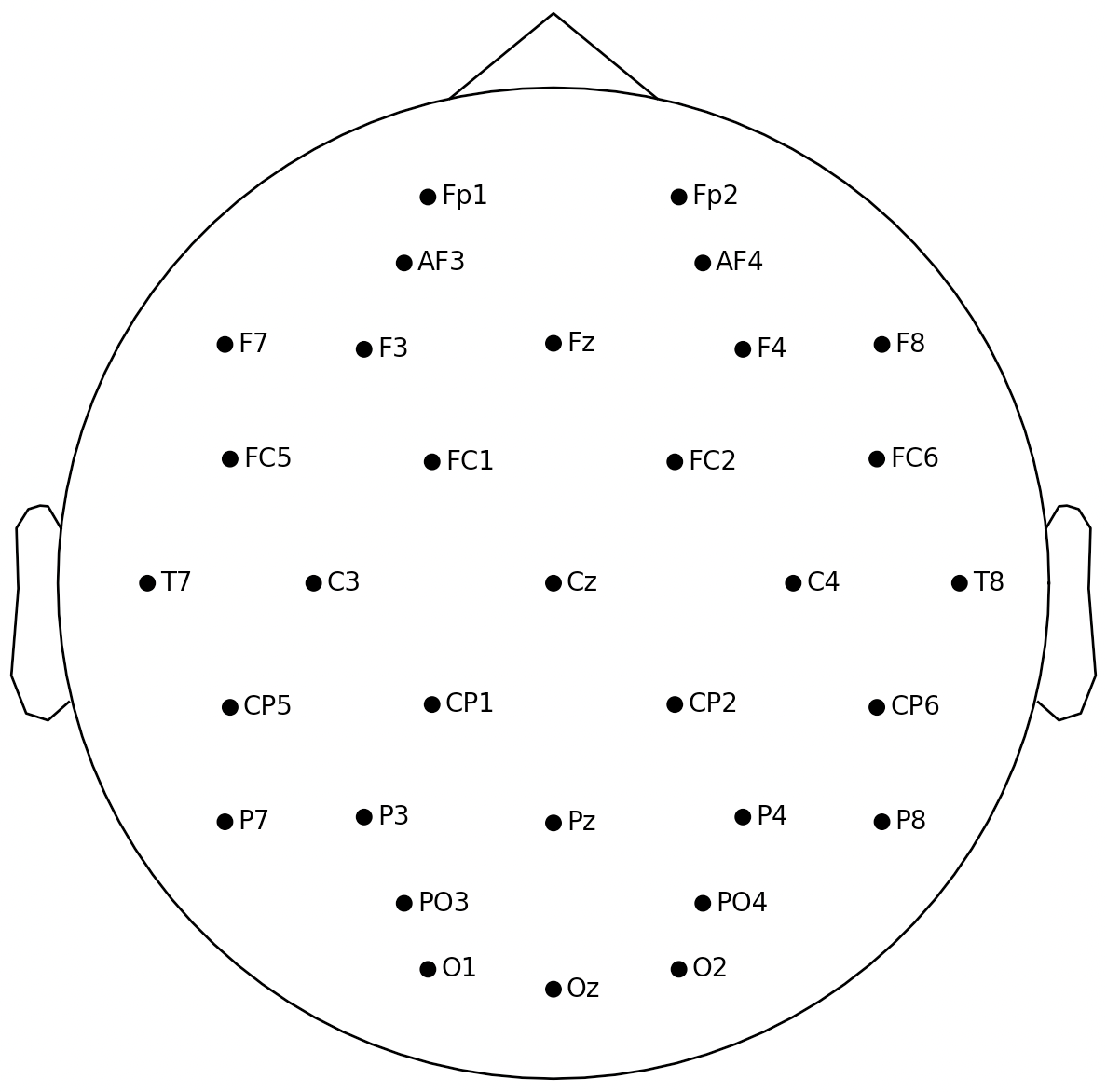
2.2 构建自定义32导联的字典,导联名称与导联权重一一对应
1
| weight = {'Fp1': 0.31, 'AF3': -0.26, 'F7': 1.22, 'F3': 0.99, 'Fz': 0.71, 'FC5': 0.55, 'FC1': -1.19, 'T7': 0.61, 'C3': -0.80, 'Cz': 2.36, 'CP5': -0.74, 'CP1': 0.72, 'P7': 0.93, 'P3': 0.38, 'Pz': 1.07, 'PO3': -1.46, 'O1': -0.12, 'Oz': 1.074, 'Fp2': 1.04, 'AF4': -0.065, 'F4': -0.52, 'F8': 0.37, 'FC2': 1.30, 'FC6': 0.94, 'C4': -1.11, 'T8': -0.16, 'CP2': 1.82, 'CP6': 0.41, 'P4': 0.46, 'P8': 0.99, 'PO4':0.15, 'O2':0.23}
|
2.3 根据脑地形图导联顺序重构自定义矩阵(*注意)
1
2
3
4
5
6
7
8
9
10
11
| # 查看脑地形图矩阵导联位置
sensor_data_32 = int32_montage.get_positions()['ch_pos']
sensor_dataframe_32 = pd.DataFrame(sensor_data_32).T
chLa_index = sensor_dataframe_32.index.values
print(chLa_index)
# 重构自定义矩阵顺序
reWeight = []
for key in chLa_index:
val = weight[key]
reWeight.append(val)
|
2.4 TopoMap可视化
2.4.1 创建info
1
2
3
4
5
| info = mne.create_info(
ch_names = chLa_index,
ch_types = ['eeg']*32, # 通道个数
sfreq = 1000) # 采样频率
info.set_montage(int32_montage)
|
2.4.2 可视化
1
2
3
4
5
6
7
| im, cn = mne.viz.plot_topomap(reWeight,
info,
names = chLa_index.tolist(),
# vlim=(-2, 2)
)
plt.colorbar(im)
plt.show()
|
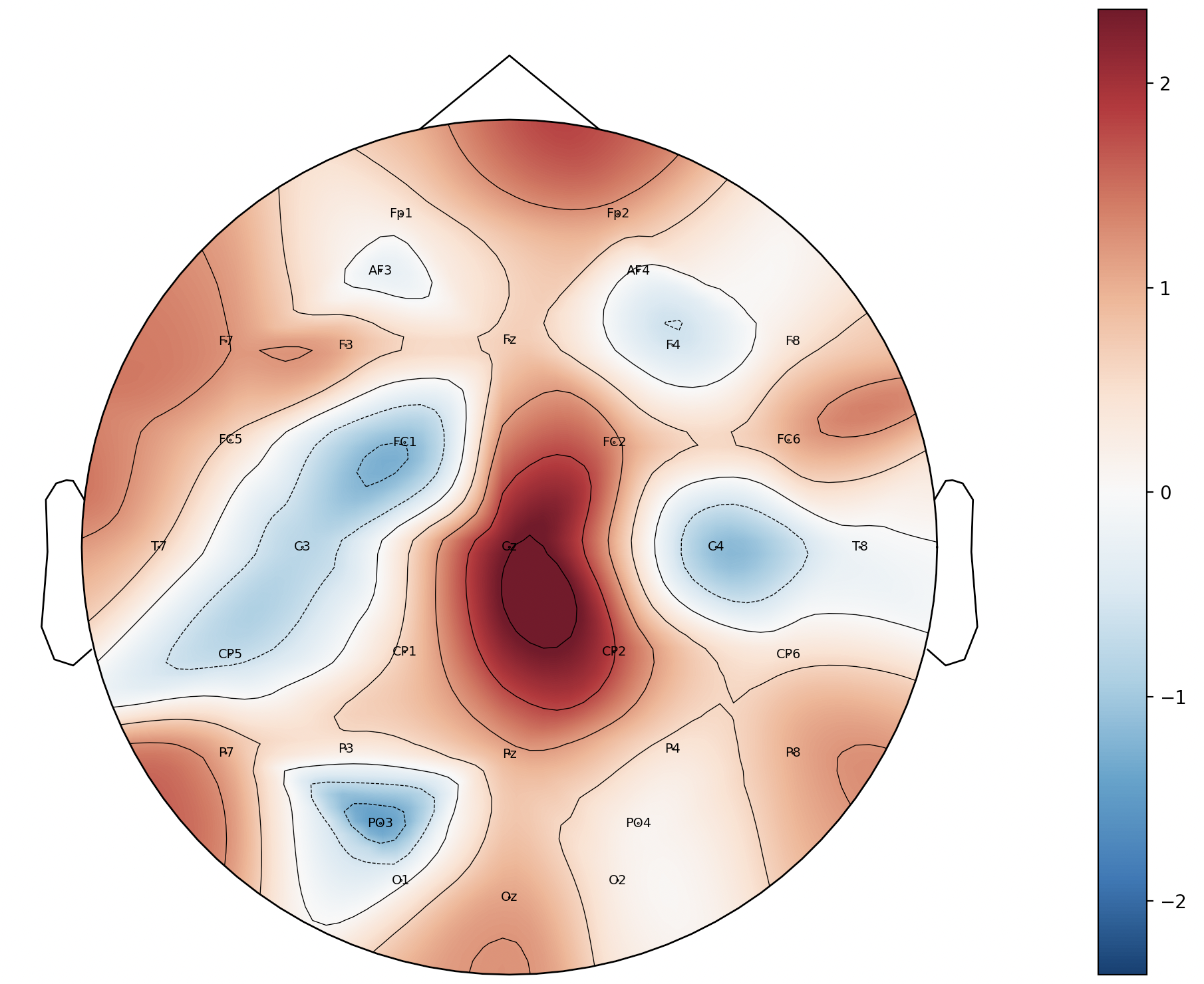
3、自定义电极布局文件对矩阵进行通道定位
假设我有一个64导联的帽子,根据实验需求,实验过程中我只选择中央区和顶叶区域的电极。
那么我们该如何根据自己选择的导联制定通道定位模版呢?
这里有两种方法:
第一种:自定义新的电极布局文件,文件只包含实验选择的导联。这个方法的目的是教会你如何制作自己的电极布局文件,实际绘制时建议选择第二种方法。
第二种:使用原来的电极布局文件,先对矩阵进行归一化,后将未被选择的导联值置为0
4、第一种方法
4.1 下载标准的64通道电极布局系统坐标
1
2
3
4
5
| # 读取MNE中biosemi64电极位置信息
biosemi_montage = mne.channels.make_standard_montage('biosemi64')
sensor_data_64 = biosemi_montage.get_positions()['ch_pos']
sensor_dataframe_64 = pd.DataFrame(sensor_data_64).T
sensor_dataframe_64.to_excel('sensor_dataframe_64.xlsx')
|
4.2 根据下载文件,选择实验选取的电极坐标,保存为excel格式
1
| # 模仿下载的电极布局excel,自定义电极布局文件
|
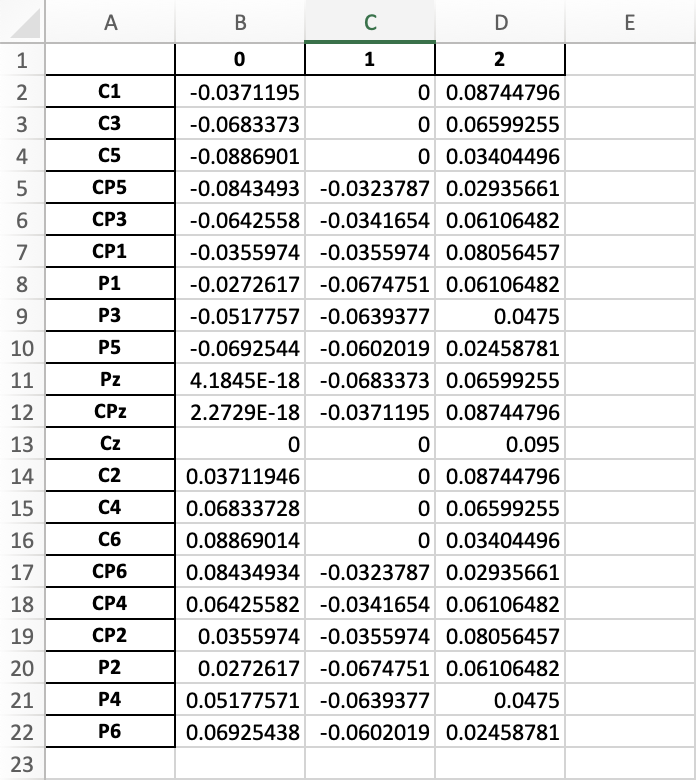
4.3 加载自定义的excel文件,制作自己的montage
1
2
3
4
5
6
7
| myStardart = pd.read_excel('mySensor_dataframe.xlsx', index_col=0) # 读取自己的电极定位文件
ch_names = np.array(myStardart.index) # 电极名称
position = np.array(myStardart) # 电极坐标位置
sensorPosition = dict(zip(ch_names, position)) # 制定为字典的形式
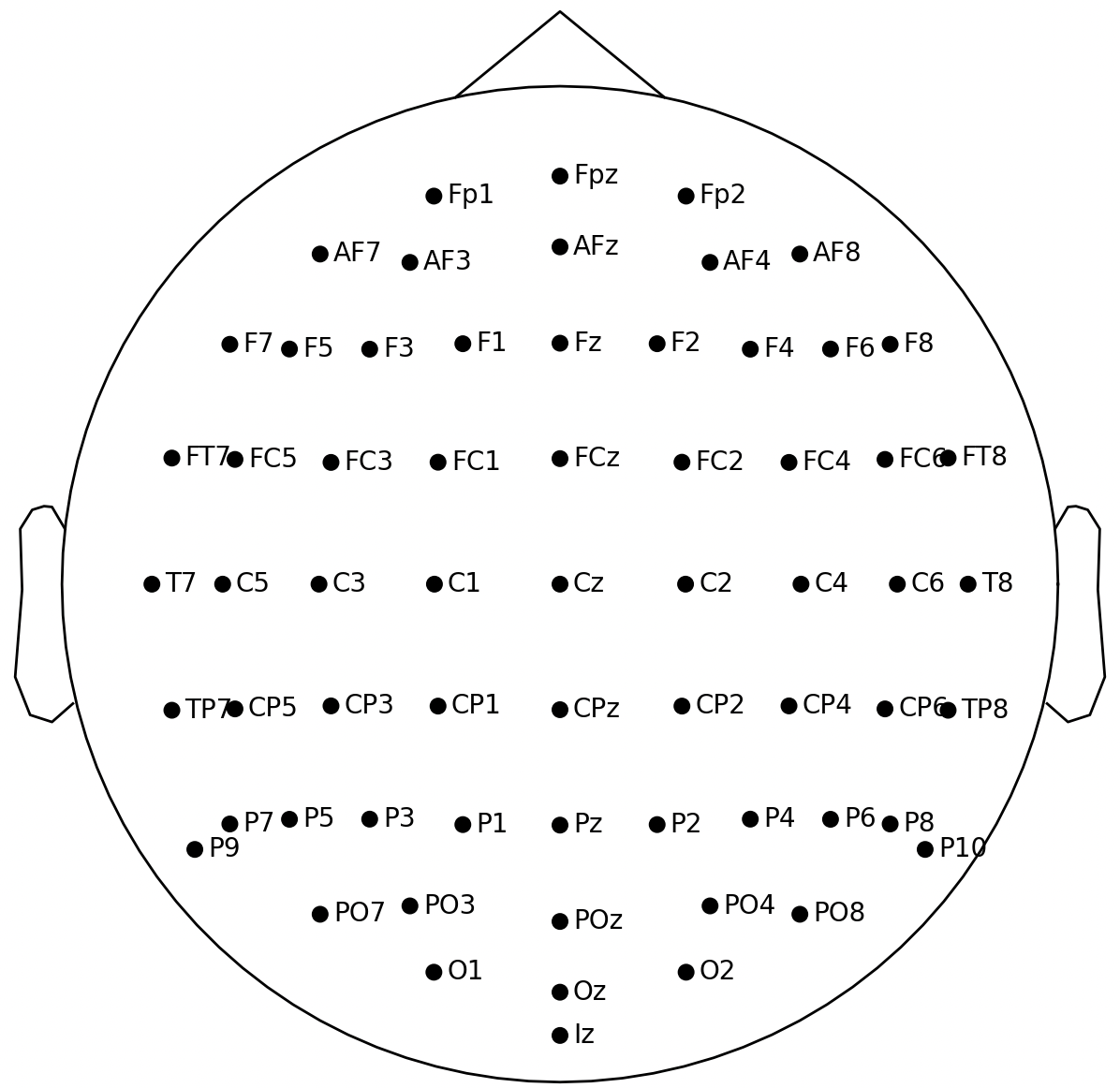
myMontage = mne.channels.make_dig_montage(ch_pos=sensorPosition)
myMontage.plot()
plt.show()
|
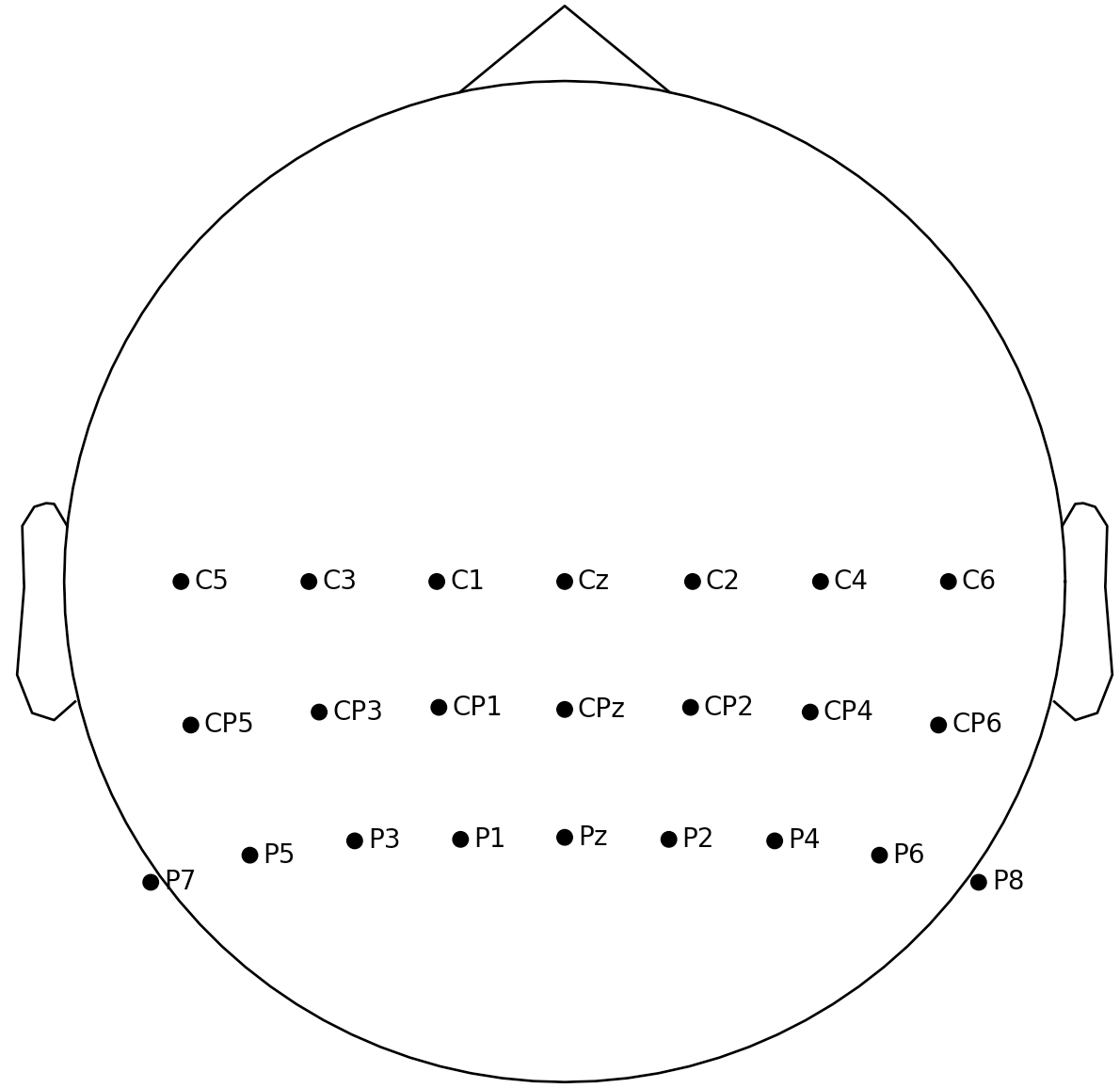
4.4 自定义导联权重字典
1
2
3
4
| myWeight = {'C1': 0.31, 'C3': 1.22, 'P6': 0.71, 'P4': 0.55,'P2': -1.19, 'CP5': 0.61,
'CP3': -0.80, 'CP1': 2.36, 'P1': -0.74, 'P3': 0.72,'P5': 0.93, 'Pz': -1.46,
'CPz': -0.12, 'Cz': 1.074,'C5':2.34,'C2': 1.04, 'C4': -0.065, 'C6': -0.52,
'CP2': 0.37, 'CP4': 1.30, 'CP6': 0.94}
|
4.5 根据脑地形图导联顺序重构自定义矩阵(*注意)
1
2
3
4
5
6
7
8
9
| # 查看脑地形图矩阵导联位置
my_chLa_index = ch_names.tolist()
print('脑地形图矩阵导联顺序:',my_chLa_index)
# 重构自定义矩阵顺序
reMyWeight = []
for key in my_chLa_index:
val = myWeight[key]
reMyWeight.append(val)
|
4.6 TopoMap可视化
4.6.1 创建info
1
2
3
4
5
| myinfo = mne.create_info(
ch_names = my_chLa_index,
ch_types = ['eeg']*21, # 通道个数
sfreq = 1000) # 采样频率
myinfo.set_montage(myMontage)
|
4.6.2 可视化
1
2
3
4
5
6
7
8
| im, cn = mne.viz.plot_topomap(reMyWeight,
myinfo,
names = my_chLa_index,
# vlim=(-2, 2)
)
plt.colorbar(im)
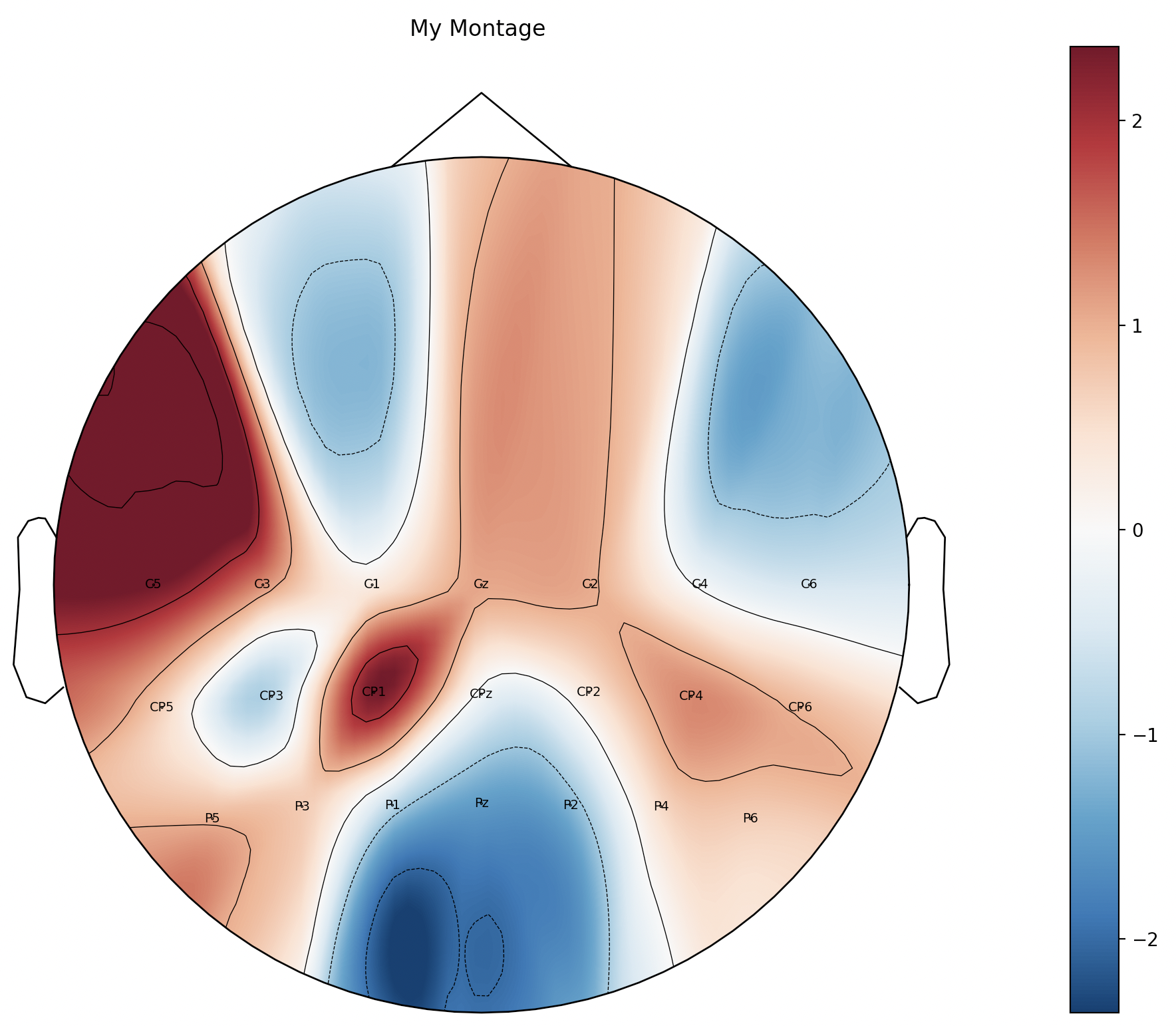
plt.title('My Montage')
plt.show()
|
5、第二种方法
5.1 读取标准的64通道电极布局系统坐标
1
2
3
4
5
6
7
| # 读取MNE中biosemi64电极位置信息
biosemi_montage = mne.channels.make_standard_montage('biosemi64')
sensor_data_64 = biosemi_montage.get_positions()['ch_pos']
sensor_dataframe_64 = pd.DataFrame(sensor_data_64).T
chLa_index_64 = sensor_dataframe_64.index.values
biosemi_montage.plot()
plt.show()
|
5.2 构建自定义部分导联字典
1
2
3
4
| myWeight = {'C1': 0.31, 'C3': 1.22, 'P6': 0.71, 'P4': 0.55,'P2': -1.19, 'CP5': 0.61,
'CP3': -0.80, 'CP1': 2.36, 'P1': -0.74, 'P3': 0.72,'P5': 0.93, 'Pz': -1.46,
'CPz': -0.12, 'Cz': 1.074,'C5':2.34,'C2': 1.04, 'C4': -0.065, 'C6': -0.52,
'CP2': 0.37, 'CP4': 1.30, 'CP6': 0.94}
|
5.3 对字典的值进行归一化
1
2
3
4
5
6
7
8
9
10
11
12
| norMyWeiht = myWeight.copy()
# Step 1: 获取需要归一化的值
values = [v for v in norMyWeiht.values()]
# Step 2: 找到最小值和最大值
min_value = min(values)
max_value = max(values)
# Step 3: 对值进行归一化计算
for key, value in norMyWeiht.items():
normalized_value = (value - min_value) / (max_value - min_value)
norMyWeiht[key] = normalized_value
|
5.4 根据脑地形图导联顺序重构标准化后的矩阵,将未被选择的导联值置为0
1
2
3
4
5
6
7
8
9
| print('脑地形图矩阵导联顺序:',chLa_index_64)
# 重构自定义矩阵顺序
reNorMyWeight = []
for key in chLa_index_64:
if key in norMyWeiht:
val = norMyWeiht[key]
reNorMyWeight.append(val)
else:
reNorMyWeight.append(0)
|
5.5 TopoMap可视化
5.5.1 创建info
1
2
3
4
5
| info = mne.create_info(
ch_names = chLa_index_64.tolist(),
ch_types = ['eeg']*len(reNorMyWeight), # 通道个数
sfreq = 1000) # 采样频率
info.set_montage(biosemi_montage)
|
5.5.2 可视化
1
2
3
4
5
6
7
8
9
| im, cn = mne.viz.plot_topomap(reNorMyWeight,
info,
names = chLa_index_64,
cmap = 'jet'
# vlim=(-2, 2)
)
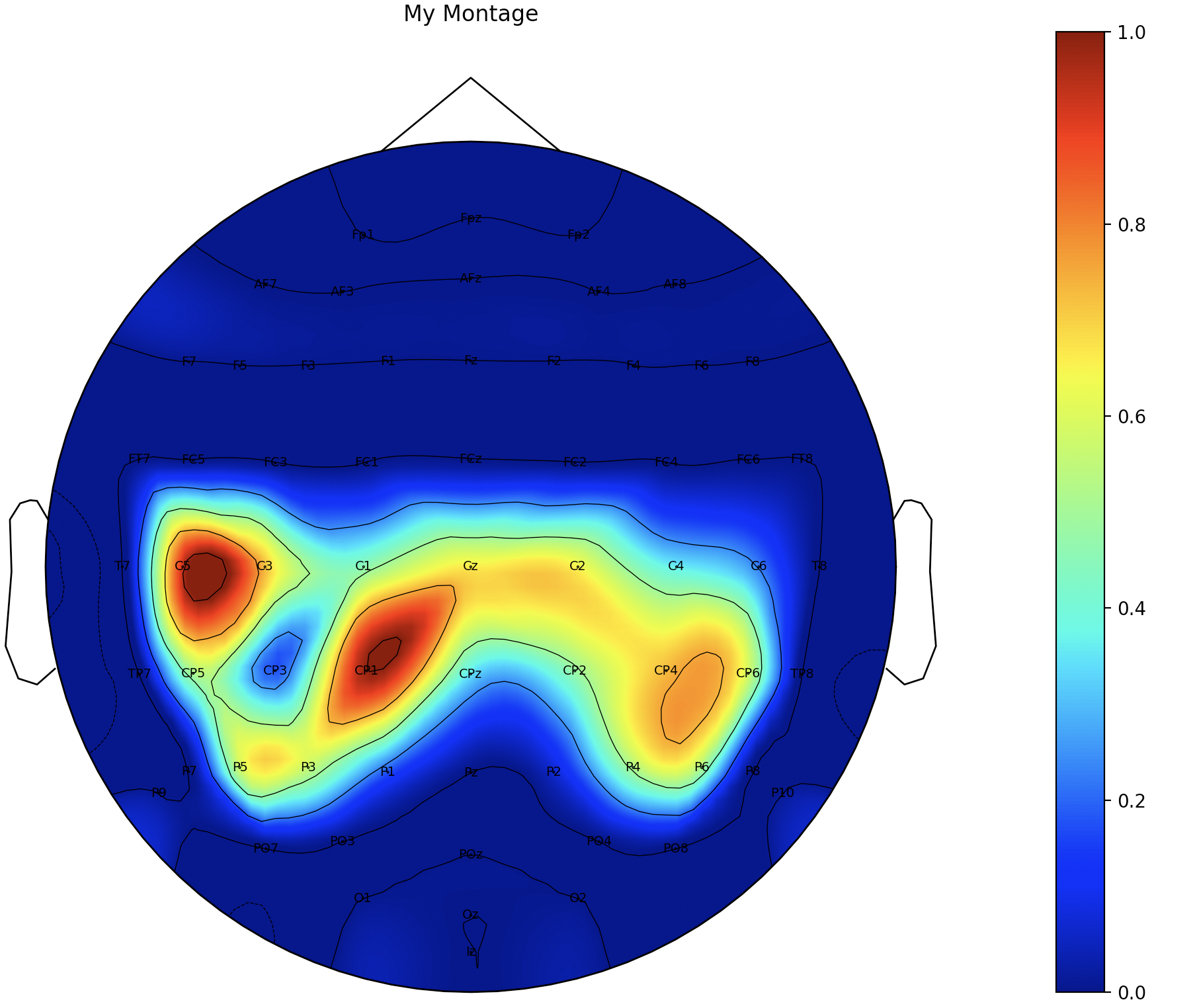
plt.colorbar(im)
plt.title('My Montage')
plt.show()
|
6、代码下载
https://github.com/weisihong9/EEG_TopoMap.git









